Escalar procesos de R usando Pachyderm
Pachyderm puede ser un alivio para procesar grandes cantidades de datos, y que mejor que utilizar R como nuestro principal motor para procesamiento y análisis de datos. Pero, ¿cómo implemento ambos para llevar un proceso a escala? ¿es esto posible?
Acompañamos en iniciativas y proyectos de ciencia de datos, ingeniería e infraestructura. Visita nuestra página ixpantia y contáctanos.
Introducción
Cuando llegué a ixpantia nunca me hice preguntas acerca de como llevar un proceso a escala, porque quizás nunca me ví en una situación donde tenía que manejar 232 GB (gigabytes) de archivos de datos. Mi experiencia en análisis de datos fuera de ixpantia se limitaba a manejar archivos de datos en formatos como .xlsx, .sav, .csv y algunos otros que nosotros los estadísticos frecuentamos manejar. Además estos archivos generalmente tenían poco peso.
Cuando llegué aquí, aprendí que una forma para agilizar el manejo de datos a mayor volumen es ponerlos en una base de datos, en donde es mucho más rápido consultar y procesar grandes cantidades de datos, así como traerlos preprocesados a una sesión de R. Pero eso me limita a escribir código en dplyr que se puede traducir al SQL de PostgreSQL. Hay muchos casos donde eso no es suficiente y debemos ir más allá de lo que podemos hacer en SQL (si no han trabajado dplyr contra una base de datos: para aplicar algunas funciones fuera de dplyr, nos obliga a hacer un collect()).
En un punto de mi historia, me enfrenté a una situación donde tenía 232 GB de datos. El pre-procesamiento necesario consistía en ejecutar un código de R bastante complejo que disminuiría considerablemente la cantidad de datos, por lo que posteriormente podría trabajarlos en R sin ningún problema. Sin embargo, para procesar solo 3 GB de datos el servidor donde estaba se demoraba más de cinco horas. Si han estado aquí posiblemente se identifiquen conmigo.
Pachyderm
Una solución pragmática es dividir los datos en varios subconjuntos, levantar múltiples máquinas virtuales y correr el script en R para cada uno de estos subconjuntos. Esto mismo se podría hacer levantando el proceso en varios contenedores Docker, que se levantan sobre un cluster de computadoras corriendo Kubernetes. ¿Pero que tal si pueden hacer eso usando una herramienta hecha específicamente para trabajar con datos sobre infraestructura de cómputo distribuida?
Aquí es donde entra Pachyderm.

Pachyderm se puede ver como un sistema de control de versiones para datos. Lo mejor de todo es que no solo crea versiones de nuestros datos, sino que también nos permite transformarlos y mantenerlos actualizados de manera eficiente a través de un dataducto (data pipeline), en donde cada vez que entran nuevos datos, Pachyderm se encarga de procesar únicamente estos datos nuevos por medio de este dataducto.
Escalar con Pachyderm
Algo que en lo personal me gusta mucho de Pachyderm es que fue creado para que los científicos de datos se concentren en ciencia de datos y no en infraestructura. Pachyderm fue implementado para trabajar bajo línea de comandos, pero con una sintaxis sumamente intuitiva. Algo que ayuda es que el sistema de control de versiones Git también se puede trabajar en línea de comandos, por lo que para los científicos de datos que utilizan Git en su día a día, puede que Pachyderm no sea muy complejo de utilizar. ¡Son muy similares!
A pesar de esta similitud, existen algunas diferencias entre Git y Pachyderm, la principal es que Pachyderm almacena archivos de datos binarios y grandes conjuntos de datos que no se procesan de la misma manera que lo hace Git. El repositorio de Pachyderm, por ejemplo, no funciona con una copia local en nuestro computador, pues todo se mantiene remotamente sobre el cluster. Pero Pachyderm sí comparte el concepto de cambios (commmits) con Git y nosotros como usuarios podemos guardar versiones de los datos en cualquier punto de la historia y cada cambio (commit) tiene su id único, al igual que en Git.
Procesar datos con Pachyderm
Para procesar datos tenemos el repositorio de entrada y el repositorio de salida que se actualizan a través de un pipeline. Este pipeline especifica las acciones que se deben realizar para procesar los datos y algunas especificaciones de cómo deben hacerse estas acciones. Pachyderm tiene una unidad mínima de datos que procesa, y a estos se les llama datum. El datum lo definimos nosotros. Por ejemplo, cada datum puede ser una de miles de fotografias, o una carpeta con archivos json, entre miles de carpetas con contenido similar. La manera en que los definimos depende de lo que queremos que Pachyderm procese cada vez que se ingresan nuevos datos y el interés de análisis en particular.
Pero bueno, ¿cómo hice yo para utilizar mi código de R para procesar mis 232 GB de datos? La respuesta no es tan simple, pero lo que sí es simple es darse cuenta que Pachyderm es una maravilla. Para resumir mi historia lo que hice en principio fue plantear mi código en R tal y como lo haría para cualquier análisis, en donde el objetivo de mi código en este caso era pre-procesar unos datos muy complejos y prepararlos para hacer un análisis estadístico específico. Mi script lo construí utilizando una pequeña parte de estos 232 GB (aproximadamente 2.5 GB).
Después de tener el script de pre-procesamiento funcionando, tenía la tarea de escalarlo. Sin embargo, cuando llegué a Pachyderm no era muy claro por donde debía comenzar, a pesar de que la documentación es bastante completa. El primero de los retos fue poder usar Pachyderm en Windows, pues tuve que utilizar WSL (Windows Subsystem for Linux) para poder correrlo. Esto involucró instalar Ubuntu desde la Microsoft Store, y posteriormente trabajar minikube desde el Power Shell de Windows y Pachyderm desde WSL. Fue un poco frustrante porque cada vez que arrancaba una máquina en minikube tenía que actualizar a la misma IP en Pachyderm y en Kubernetes que estaban del lado de WSL. (Nota: si desean evitar todo esto pueden arrancar un entorno de prueba en el Pachyderm Hub para experimentar).
Una vez que Pachyderm estaba funcionando, lo que hice fue levantar un contenedor en Docker. Dentro de este contenedor instalé R y le ordené que cada vez que levantaba la imagen se ejecutara mi script en R. Eso si, para que Pachyderm pueda trabajar con esta imagen en Minikube, es importanten leer la documentación para usar el registro de contenedores (container registry) de Minikube. De otra forma no es accesible para tu cluster kubernetes local.
Con la imagen lista el siguiente paso es especificar los parámetros en el dataducto (pipeline) que es donde le ordenamos a Pachyderm qué debe hacer. En mi caso debía tomar la imagen del contenedor y ejecutar mi script de R con cada uno de los datum, que se almacenan en el repositorio de entrada. Una vez procesado cada datum Pachyderm va depositando los datos preprocesados en el repositorio de salida y no queda más que sacarlos de ahí.
Kubernetes
Kubernetes es un sistema de código libre diseñado por Google que sirve para la automatización, ajuste de escala y manejo de aplicaciones en contenedores; aquí la unidad mínima de trabajo es un pod, entonces lo que hace Kubernetes es orquestar sus pods para que funcionen de la manera más eficaz posible sobre los recursos de computo disponibles. La idea principal para procesar mis 232 GB con Pachyderm era levantar un cluster de Kubernetes en Google Cloud Platform (a través de Google Kubernetes Engine), dónde se ejecutaría el proceso en R de la siguiente manera:
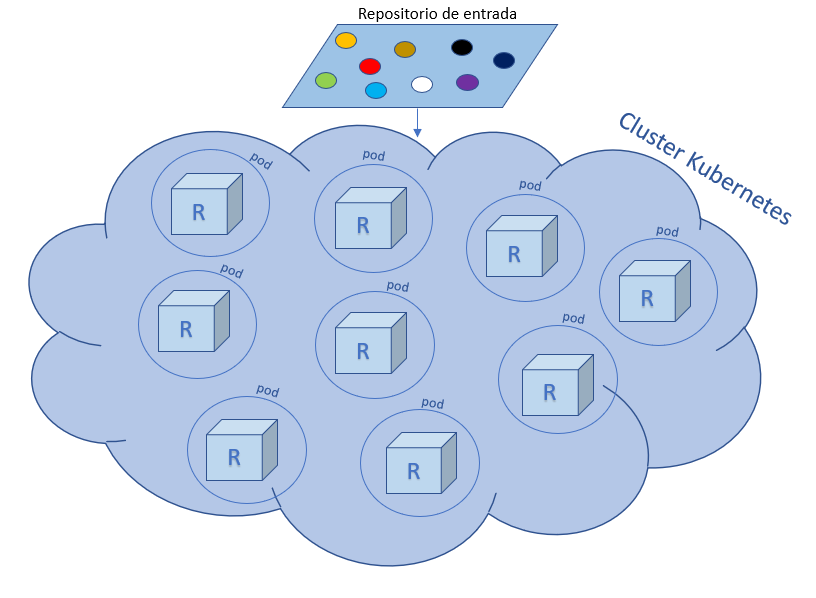
En la imagen de arriba se observan varios pods trabajando dentro del cluster, y en cada pod ponemos un contenedor que es como una pequeña máquina virtual con R instalado para ejecutar nuestro código. Una vez que tenemos esta infraestructura lista, Pachyderm se encargará de llevar cada uno de los datum, en nuestro repositorio de entrada, a cada uno de estos pods para procesarlos.
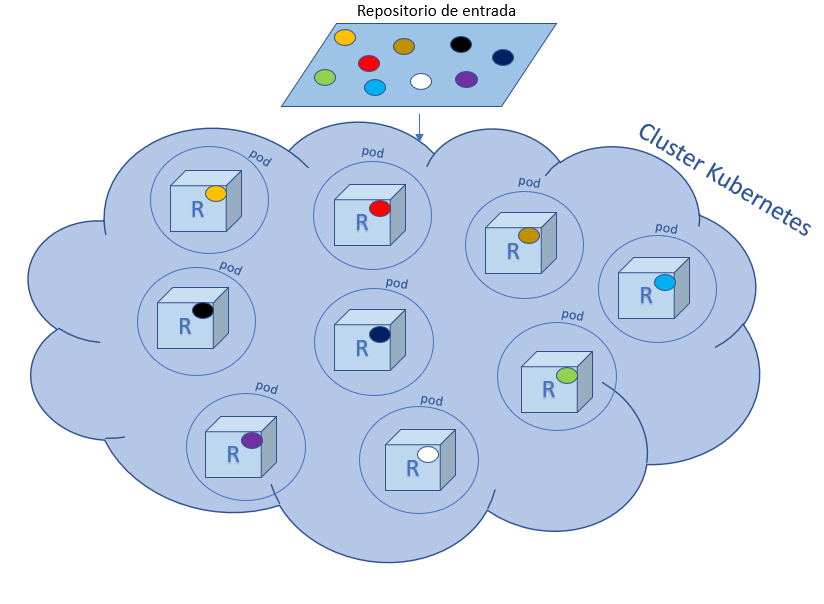
Cada pod trabaja con un solo datum. Cada datum es procesado con R y una vez procesado se envía al repositorio de salida de Pachyderm: datos recien horneados y listos para el siguiente paso de analisis… ¡justo lo que necesitaba!
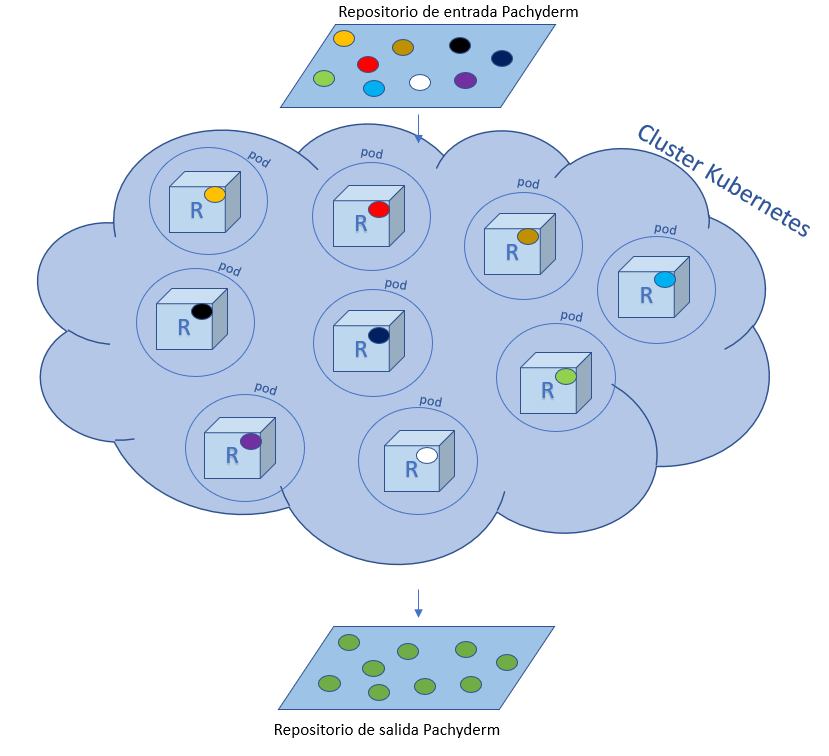
En la definición del pipeline que usamos en Pachyderm, podemos especificar diversos parámetros, entre ellos limitar la memoria que se le asigna a cada pod y la cantidad de núcleos que usa cada pod. En mi caso aumentamos el tamaño del cluster para tener más máquinas trabajando y horneando datos. Lo que al principio fue un proceso que llevaba hasta 24 horas, lo logramos correr en 45 minutos utilizando 10 servidores de 8 CPU con 52 GB de memoria (en total 80 CPU y 520 GB de memoria en el cluster). Y aunque suena extravagante, ¡correr un cluster de este tamaño cuesta menos de USD 10 por hora!
Análisis reproducible
Lo bueno de Pachyderm es que al igual que en R tenemos código y lo podemos entregar como un proceso reproducible para nuestros clientes. No es algo que implementemos una vez y el procedimiento quedó perdido; esto cumple con el principio de reproducibilidad en que tanto se insiste en la comunidad científica. Además en ixpantia no creemos en las cajas negras. Por el contrario siempre buscamos dejar todo en manos del cliente para que este pueda repetir o en dado caso adaptar el proceso a nuevas necesidades.
Después de todo este proceso finalmente logré entender la imagen que puse de portada:

A veces debemos hacer muchos malabares sobre nuestros datos para poder llegar a un análisis concreto, al igual que lo hace este elefante sobre sus cubitos de datos. Ahora que me considero un elefante puedo decir que fue toda una aventura usar Pachyderm por primera vez.
Desde mi punto de vista le recomendaría usar Pachyderm a cualquier persona que desea llevar su código de R a escalas realmente masivas. En la documentación de Pachyderm hay varios ejemplos que se pueden seguir paso a paso; esto en caso de que este relato los haya inspirado a experimentar más sobre el tema.
Este blog lo mantiene el equipo de ixpantia y la comunidad de gente interesada en datos de la cual estamos contentos de formar parte ¿Tienes una idea para publicar algo aquí? ¡Escríbenos! Estamos siempre interesados en material e ideas nuevas. © 2019-2022 ixpantia